
Google搜索了一下,发现隐式方程的求参问题有不少解决方案,matlab, R都可以实现。基本思路与我用Excel求参的方法一样,先编写一个函数来实现隐式方程的显示化,matlab中要用到fzero或fsolve(Excel中用的是单变量求解插件),然后再用非线性求解函数进行参数拟合。下面是积分后的米曼方程(IMM)的matlab求参代码:
先构造IMM函数:
function y=IMM(p,x)
%输入参数
Vm=p(1);
Km=p(2);
y=zeros(size(x));
NN=length(x);
%不输出提示
opt = optimset('display','off');
for i=1:NN
%就是用这个方程来求出某个x(i)下的解,y的初值设定为x(i)*0.9
y(i)=fzero(@(y) x(i) -y-Vm*15+Km*log(x(i)/y), x(i)*0.9, opt);
end
end
上面的函数写好后,保存,最后只要用下面一行命令就可以求解参数了:
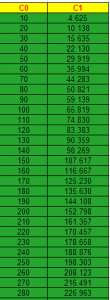
X=lsqcurvefit(@(params, xdata) IMM(params, xdata),... [10 10], C0, C1) %[10,10]分别是Vm和Km的初始值,C0和C1为起始浓度和反应15分钟后的浓度, %求到的参数存在X中,第一个为Vm,第二个为Km
lsqcurvefit支持”trust region reflective”和“Levenberg-Marquardt”两种非线性拟合算法,可通过OPTIONS = optimset(‘Algorithm’,’levenberg-marquardt’)指定。我用加了SD=1的噪音的数据(见下图)进行验证,求到的参数与理论值[5, 10]很接近,为[5.0109, 101.3801,Excel求的结果为[4.9649, 99.0731]。这些差异可能与不同算法的收敛标准或者对数值的取舍精度不同造成的,貌似matlab数据处理的精度更高一些。
–EOF–
文章来自[MS: 质谱与建模],微信号:MS4Fun,不定期发布自己在质谱应用和建模&模拟方面遇到的一些有趣的事情,欢迎分享与推荐。
